近日,我校yl23455永利王海峰教授团队成功发布了中国大豆品种“中黄13”(Zh23)的完整基因组“Zh23-T2T”,并首次绘制了该大豆品种的表观遗传修饰图谱。这一研究结果以 “The T2T genome assembly of soybean cultivar Zh23 and its epigenetic landscapes” 为题发表于《分子植物》(Molecular Plant)。广西大学是第一署名单位,广西大学博士生张超、助理教授谢亮、博士生于航以及东北农业大学副教授王锦辉为论文的共同第一作者,广西大学王海峰教授和东北农业大学陈庆山教授为该论文的通讯作者。

《分子植物》(Molecular Plant)是Cell Press出版社旗下植物学领域最高层期刊之一,是中国科学院JCR分区一区,最新影响因子27.5(2022年)。
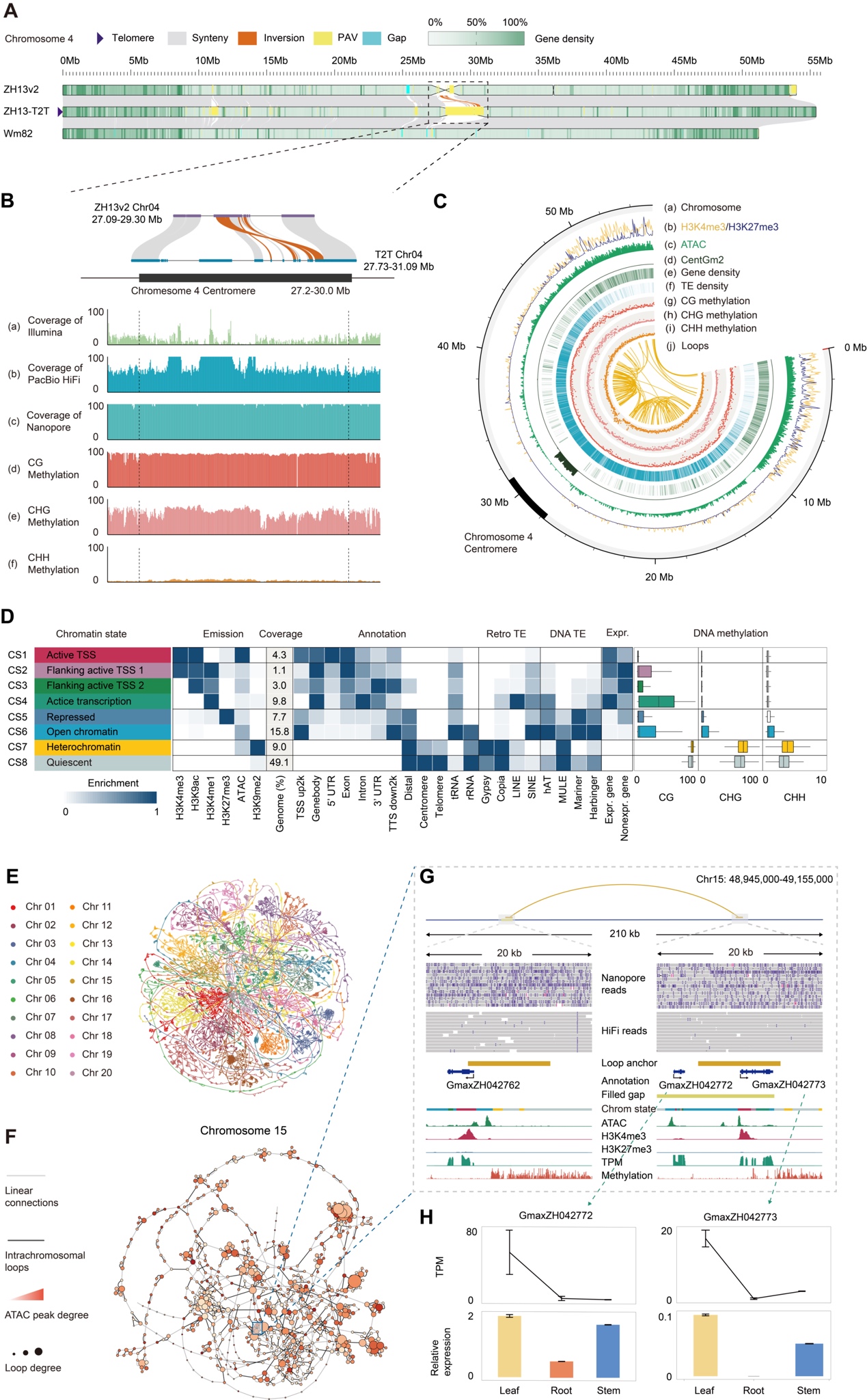
研究人员首先整合了Nanopore超长测序、PacBio HiFi测序和Hi-C技术,成功组装了近乎完整的Zh23-T2T基因组(图A),填补了Zh23v2基因组的空白区域,特别是着丝粒和端粒区域。作者补充了约26Mb的新序列,并校正了35个倒位和163个易位,其中大部分结构变异发生在着丝粒区域(图B),为进一步研究大豆的功能基因组提供了坚实的数据基础。
为了探讨大豆Zh23-T2T基因组的表观修饰模式,研究人员整合了多种组蛋白修饰测序数据,包括H3K4me3、H3K27me3、H3K9ac、H3K4me1和H3K9me2,以及染色质开放水平测序数据(ATAC-seq),将基因组分为8种不同的染色质状态。这一工作揭示了具有不同生化特性的各种基因组区域的分布,并突显了不同染色质状态下DNA甲基化、转座子(TE)富集和基因表达之间的复杂关系(见图C、D)。作者还使用Nanopore数据检测了Zh23着丝粒和端粒区域的DNA甲基化水平,并表明了第三代测序在准确捕捉复杂区域甲基化信号方面的潜力。此外,研究人员还通过分析Hi-C数据构建了Zh23的三维互作网络(图E-H),鉴定了潜在的近端和远端的调控元件,为深入研究大豆的功能元件提供了新资源。
近年来,王海峰教授团队致力于利用生物信息学手段开展植物基因组学和表观基因组学研究,解析了多种亚热带经济作物的基因组和表观基因组图谱,揭示了表观遗传修饰在基因组多倍化和多拷贝基因功能分化的重要调控作用,对利用基因组学和表观基因组学开展农作物基因功能研究和分子育种具有重要的意义。系列研究成果在PNAS、Molecular Plant、Plant Biotechnology Journal、Plant Journal等国际知名期刊发表SCI论文30余篇,累计引用1700余次。

“植物表观基因组学”课题组合影(前排中间左一为王海峰教授)
